SnpEff自建注释库及HGVS命名
最开始用SnpEff一直使用最近SnpEff提供的注释库,通过snpEff命令即可查找所需要的数据库名称
java -jar snpEff.jar databases |less -S
而且SnpEff官网也说明只用其默认的提供的数据库即可
SnpEff databases for the most popular genomes are already pre-built and available for you to download. So, chances are that you don’t need to build a database yourself (this will save you a LOT of work)
然后通过 download 命令下载其官网上的数据库即可
java -jar snpEff.jar download -v GRCh38.86
但是最近发现如果我们在Call variant时使用的是Ensembl最新的数据库,比如release-94版本;而我们下载的SnpEff数据库只有release-86版本(GRCh38.86),那么最终的突变注释则还是基于86版本的信息,最简单的偏差的有:
- 定位到的基因ID不同(由于一些基因ID在最新的注释版本中被移除或者新增了)
- 定位到的基因symbol发生变化(正常注释版本更新导致的)
- 定位到的转录本ID不同,如果只是ID变了,对应序列不变的话还好,如果转录本序列也变了,从而会导致HGVS.c/HGVS.p也发生变化
- 另外还有些突变位点位于两个基因之间,有时snpEff自建注释库和官网下载的注释库,对于这些位点到底属于哪个基因的选择会有点不同(我暂时也不知道是什么原因)
- 其他影响暂时还没发现
所以我觉得还是有必要将在call variant时选用的参考基因组版本与用SnpEff注释时用的基因组版本保持一致
如何建立snpEFF注释库,官网上有个针对人物种的详细例子,我以release-94版本的小鼠为例:
修改配置文件 vim snpEff.config ,在最后一行后增加对应基因组信息
# ENSEMBL release 94 Mus_musculus_94.genome : Mus_musculus_94
在snpEff目录下的data文件夹内新建Mus_musculus_94目录,用于放置snpEff库文件
mkdir Mus_musculus_94 && cd Mus_musculus_94
下载snpEff建库所需文件,并更改为固定文件名,文件分别有:
-
参考基因组序列
wget ftp://ftp.ensembl.org/pub/release-94/fasta/mus_musculus/dna/Mus_musculus.GRCm38.dna.primary_assembly.fa.gz
-
参考注释文件GTF
wget ftp://ftp.ensembl.org/pub/release-94/gtf/mus_musculus/Mus_musculus.GRCm38.94.chr.gtf.gz
-
参考蛋白序列
wget ftp://ftp.ensembl.org/pub/release-94/fasta/mus_musculus/pep/Mus_musculus.GRCm38.pep.all.fa.gz
-
参考cds序列
wget ftp://ftp.ensembl.org/pub/release-94/fasta/mus_musculus/cds/Mus_musculus.GRCm38.cds.all.fa.gz
-
参考regulatory annotations
wget ftp://ftp.ensembl.org/pub/release-94/regulation/mus_musculus/mus_musculus.GRCm38.Regulatory_Build.regulatory_features.20180516.gff.gz
解压缩后并更改名称
gunzip ./* mv Mus_musculus.GRCm38.dna.primary_assembly.fa sequences.fa mv Mus_musculus.GRCm38.93.chr.gtf genes.gtf mv Mus_musculus.GRCm38.pep.all.fa protein.fa mv Mus_musculus.GRCm38.cds.all.fa cds.fa mv mus_musculus.GRCm38.Regulatory_Build.regulatory_features.20180516.gff regulation.gff
最后先切换到snpEff目录下,然后用 snpEff.jar build一下
java -jar snpEff.jar build -gtf22 -v Mus_musculus_94
但是如果按照上述步骤来的话,会发现在 build 过程中会出现一点错误,snpEff会check protein/cds id是否跟GTF注释文件保持一致,但是现在最新的Ensembl版本下载下来的protein/cds序列中的ID都带有版本号,当在check protein id时会提示:
Protein check: Mus_musculus_94 OK: 0 Not found: 65328
也就是说SnpEff会认为65328个蛋白ID都没有匹配上,但是似乎这个不影响一般的常规VCF注释(因为其突变位点对氨基酸的影响是基于cds序列来的)
如果为了让protein序列的check能正常通过,其实只需要将protein序列中的ID后面的版本号去掉即可
HGVS(HumanGenome Variation Society)基因突变命名规则:
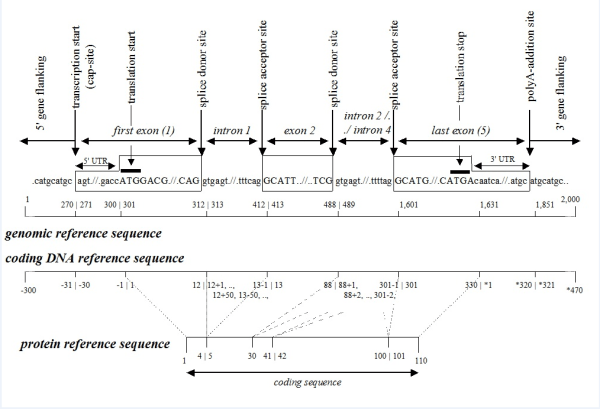
g:基因组参考序列;c:cDNA序列;p:蛋白参考序列;n:非编码DNA参考序列;r:RNA参考序列;m:线粒体DNA参考序列
核苷酸水平:
- c.-352G>C:表示cDNA上发生替换(substitution),即序列中的中某一个碱基被另外一个碱基所替代,分为转换(transition:嘌呤与嘌呤之间的替换,或者嘧啶与嘧啶之间的替换)和颠换(transversions:嘌呤与嘧啶之间的替换)
- c.-3534delT:表示缺失(deletion),即序列中一个或多个碱基缺失,多个碱基缺失:c.-352_-348delTACAG
- c.351dupT:表示重复(duplication),即序列中一个或多个碱基拷贝插入下游序列,多碱基发生重复:c.328_329dupAA
- c.3_4insC:表示插入(insertion),即序列中一个或多个碱基发生插入,非上游序列拷贝
- NC_000022.10:g.193-199inv:表示倒置(Inversions),即序列发生与原始序列反向互补的替换
- NC_000022.10:g.42522624_42522669con42536337_42536382:表示转换(conversion),即特殊类型的缺失-插入,其中替代原始序列的核苷酸序列是来自基因组的另外一个位点的序列
- c.142_144delinsTGG:表示缺失-插入(deletion-insertion),即序列原有的一个或多个核苷被新的一个或多个核苷取代,且不是替换(substitution)、倒置(inversion)和转换(conversion)
- NC_000014.8:g.101179660TG[14]:表示重复序列(Repeat sequences),即序列出现了一个或多个核苷酸发生重复
- 对于cDNA一些内含子的位置,如:c.377+1014A>G,表示前面一个外显子的位置是在377,然后下游1014位置的碱基A替换成G
- 对于cDNA的5’UTR的位置,如:c.-9T>G,表示以第一个外显子转录开始位置(1)上游第9个碱基T替换成G
- 对于cDNA的3’UTR的位置,如:c.*9T>G,表示以最后一个外显子转录结束位置的下游第9个碱基T替换成G
蛋白水平(基本上与核苷酸水平类似):
- p.W123*:星号表示终止密码子,也可以用Ter表示
- p.Ter806Argext*?:ext表示突变对起始和终止密码子在N端或C端延伸的影响,这例子表示终止密码子变成了Arg
- p.Met1?:表示翻译起始密码子缺失,还例如:p.Met1ValextMet-12表示DNA序列上c.1A>G的变异导致最终蛋白产物在N端延伸了12个氨基酸,Met变异为Val
- p.Val972fs:表示移码突变,即972位Val是首个发生改变的氨基酸;如p.Arg97ProfsTer23,表示第97位的Arg是首个发生改变的氨基酸,且Arg变为Pro,同时发生移码突变后,终止密码的位置变为第23位
突变命名详细说明可查阅: Sequence Variant Nomenclature
参考资料:
最新HGVS基因突变命名规则速览
人类基因组变异标准命名(HGVS)本文出自于 http://www.bioinfo-scrounger.com 转载请注明出处










![[HBLOG]公众号](https://www.liuhaihua.cn/img/qrcode_gzh.jpg)

